第一作者:丁西朋(1982-),男,河南封丘人,助理研究员,博士,主要从事热带牧草遗传育种研究。E-mail:[email protected]
为建立柱花草( Stylosanthes spp.)DUS测试标准品种DNA指纹图谱数据库,实现柱花草品种的快速、准确鉴定,利用筛选到的25个SSR标记对15份柱花草DUS测试标准品种进行了遗传多样性分析及DNA指纹图谱构建。结果表明,25个SSR标记共产生132个等位基因,每个标记可产生3~9个等位基因,平均5.280个;各SSR标记的多态性信息含量为0.393~0.847,平均为0.638。利用这25个SSR标记构建的指纹图谱能有效区分15份柱花草DUS标准品种,并为每份标准品种建立了QR二维码图谱,为SSR标记技术在柱花草DUS测试中的应用提供依据。
DNA fingerprinting of Stylosanthes standard cultivars for distinctness, uniformity and stability (DUS) testing was constructed to facilitate the identification of Styosanthes cultivars. The genetic diversity analysis and DNA fingerprinting construction of 15 Stylosanthes standard cultivars for DUS testing were performed using 25 SSR markers. The results indicated that there were total of 132 alleles were generated from 25 SSR markers among these 15 standard cultivars. The number of alleles generated from each SSR marker ranged from 3~9, with an average of 5.280 alleles. The polymorphic information content per SSR marker varied from 0.393 to 0.847, with an average of 0.638. The 15 standard cultivars could be successfully distinguished by DNA fingerprinting, which was constructed based on these 25 SSR markers. The DNA fingerprinting QR code for each Stylosanthes standard cultivars was constructed. The results could provide important reference for application of SSR marker in Stylosanthes DUS testing.
随着育种业和种子贸易的发展, 植物新品种权作为知识产权的一种形式, 其重要性越来越突出。DUS测试, 即特异性(Distinctness)、一致性(Uniformity)和稳定性(Stability)测试, 是植物新品种保护的技术基础和授权的科学依据[1]。建立和完善DUS测试技术体系, 对于植物新品种权保护的发展有重要作用和现实意义。标准品种是植物新品种DUS测试中对品种性状尤其是数量性状进行客观描述的参照品种, 是DUS测试技术体系的重要组成部分, 构建标准品种完整的形态性状及指纹图谱数据库是开展植物品种DUS测试的必备数据基础[2]。
传统的DUS测试主要以植物的表观形态特征为基础, 测试结果受环境影响大、稳定性差、测试周期长, 严重地阻碍了我国植物新品种授权速度[3]。因此, 植物新品种DUS快速测试技术的研究和建立十分紧迫。以分子标记为基础的DNA指纹技术具有测试周期短、不受环境影响、易于自动化等优势, 是DUS快速测试技术的发展方向[4]。SSR(Simple Sequence Repeats)标记具有数量丰富、稳定重复性好、多态性高、共显性、操作简单等优点, 被认为是植物新品种DUS测试中较理想的标记技术, 已经成功应用于棉花(Gossypium hirsutum)[5]、油菜(Brassica napus)[6]、大麦(Hordeum valgure)[7]、西瓜(Citrullus lanatus)[8]、玉米(Zea mays)[9]、水稻(Oryza sativa)[10]、小麦(Triticum aestivum)[11]和番茄(Lycopersicon esculentum)[11]等多种农作物标准品种的指纹图谱构建及DUS测试中。
柱花草(Stylosanthes)为多年生豆科植物, 具有适应性广、耐干旱、耐酸瘠土、抗病性强、饲草产量和营养价值高等特点, 是世界上热带及亚热带地区最重要的放牧和刈割兼用型豆科牧草[12]。我国于1957年开始从国外引进柱花草进行栽培, 先后培育出维拉诺柱花草(S. hamata cv. Verano)、热研2号柱花草(S. guianensis cv. Reyan No.2)、热引18号柱花草(S. guianensis cv. Reyin No.18)、热研20号柱花草(S. guianensis cv. Reyan No.20)等多个柱花草优良品种[13, 14]。柱花草属于典型的自花授粉植物, 且在开花之前已经授粉, 异交率仅为1%~2%[15, 16]。柱花草属包含约50个种或亚种, 染色体基数为10, 大部分柱花草为二倍体植物(2n=20), 也有部分为四倍体或六倍体(2n=40或2n=60)植物[17]。2012年, 农业部发布了《植物新品种特异性、一致性和稳定性测试指南 柱花草》报批稿, 指南中提出了柱花草DUS测试的24个必测植物学性状。中国热带农业科学院热带作物品种资源研究所热带牧草研究中心通过对192份柱花草种质的植物学性状进行测试, 对该DUS测试指南提出修改建议并确定了15个标准品种[18]。随着柱花草品种的增多, 其形态特征将会越来越复杂, 使得依据形态性状进行柱花草品种鉴别越来越困难, 因此, 将SSR标记技术引入到柱花草DUS测试中十分必要。据报道, 在柱花草中可用的SSR标记共139个, 分别来自柱花草属的不同种, 其中44个来自圭亚那柱花草(S. guianensis)、26个来自大头柱花草(S. macrocephala)、23个来自灌木柱花草(S. seabrana)、23个来自头状柱花草(S. capitata), 其余来自其他14个不同的柱花草种[19, 20, 21, 22, 23, 24]。热带牧草中心热带牧草研究中心对其中123个SSR标记在8个柱花草种间的转移性进行了分析, 结果表明, 44个在8种柱花草中都能有效扩增, 其中26个多态性较好[25]。本研究在前期工作基础上, 利用25个SSR标记对15个柱花草DUS测试标准品种进行遗传多样性分析, 构建15份标准品种的DNA指纹图谱QR二维码数据库, 以期为SSR标记技术在柱花草DUS 测试中应用奠定基础。
试验材料除了《植物新品种特异性、一致性和稳定性测试指南 柱花草》中列出的圭亚那柱花草、有钩柱花草(S. hamata)、糙柱花草(S. scabra)、头状柱花草标准品种外, 还包括热带牧草中心对《植物新品种特异性、一致性和稳定性测试指南 柱花草》验证后建议新增的圭亚那柱花草、大头柱花草、马弓形柱花草(S. hippocampoides)、矮柱花草(S. humilis)以及细茎柱花草(S. gracilis)等其他标准品种[18], 共计15份柱花草标准品种(表1), 均种植在中国热带农业科学院热带作物品种资源研究所热带牧草研究中心基地, 每个小区面积为24 m2, 株距为80 cm, 行距为100 cm, 走道为1.5 m, 四周设置保护行, 每小区种植总株数为42。
| 表1 15份柱花草DUS测试标准品种 Table 1 15 Stylosanthes standard cultivars for DUS testing |
将去皮的柱花草种子在80 ℃水中浸泡5 min, 然后置于25 ℃恒温培养箱中发芽, 发芽后移至14 L塑料盆中进行营养液培养。培养1个月后, 每份标准品种随机挑选5株, 对叶片进行混合取样, 置于液氮中保存。按照笔者所在实验室采用的改良CTAB法提取柱花草总DNA[25], 并用1%琼脂糖凝胶电泳检测DNA样品。SSR标记选用笔者所在实验室已经筛选过在8个不同柱花草中多态性较好的25个标记(表2)[25], 均由上海生工生物工程股份有限公司合成。反应体系为20 μ L体系:1 μ L 50 ng· μ L-1模板, 0.8 μ L 5 μ mol· μ L-1上下游混合引物, 0.3 μ L 10 μ mol· μ L-1 dNTPs, 2 μ L 10× PCR Buffer, 0.2 μ L 5 U· μ L-1 Taq聚合酶, ddH2O补足。PCR程序为:94 ℃预变性4 min; 94 ℃变性40 s, 55 ℃退火40 s, 72 ℃延伸45 s, 35个循环; 72 ℃延伸5 min。8%聚丙烯酰胺凝胶(8 mL 30%丙烯酰胺/N, N’ -亚甲基双丙烯酰胺=29/1、300 μ L 10%过硫酸铵、30 μ L TEMED、3 mL 10× TBE、18.7 mL H2O)电泳, 银染检测。
根据每对SSR引物对每份材料的扩增产物电泳图在Excel表格中进行统计, 位点由大到小依次记录, 同一位点处, 有扩增条带的记为“ 1” , 没有扩增的就记为“ 0” 。用NTSYS-pc 2.10e软件进行非加权组平均(Unweighted Pair-group Method with Arithmetic Means, UPGMA)聚类分析, 其中Qualitative data计算遗传相似系数(Genetic Similarity, GS)[26], 并利用FreeTree软件[27]通过1 000次重抽样对聚类图进行bootstrap分析。利用POPGEN 1.32软件分析每对SSR引物等位基因频率(Allele Frequency)、等位基因数(Number of Alleles, Na)、有效等位基因数(Effective Number of Alleles, Ne)、Shannonx信息指数(Shannon’ s Information Index, I)及Nei’ s多样性指数(Nei’ s Gene Diversity, He)[28]。根据等位基因频率计算每个SSR标记的多态性信息含量(Ploymorphism Information Content, PIC), 其计算公式为:
PIC=1-
式中, n表示每个SSR标记检测到的等位基因数目, Pi、Pj表示第i、j个等位基因在供试材料中出现的频率[29]。
| 表2 所选25个SSR标记信息 Table 2 The information of 25 SSR markers used in this study |
DNA指纹图谱构建参照范建光等[8]、宋海斌等[30]图谱代码构建的方法, 采用英文字母与数字组合的方式, 其中英文字母按照已筛选出的核心引物字母编号(A、B、C、…)依次排列(表2), 组成的数字则表示各个引物在不同标准品种中对应的基因型(将SSR引物在所有标准品种中的PCR产物按照分子量由大到小依次排列, 用其数字顺序表示对应标准品种的基因型)。如西卡柱花草部分指纹图谱QR编码为A2B6, A2表示SSR4引物在西卡柱花草中的基因型为2(SSR4引物在西卡柱花草中PCR产物的分子量是所有标准品种中第二大), B6表示SSR5引物在西卡柱花草中的基因型为6(SSR5引物在西卡柱花草中PCR产物的分子量居所有标准品种中第六大)。利用25个SSR标记在西卡柱花草的基因型, 最终形成西卡柱花草DNA指纹图谱的QR编码为A2B6C4D1E6F2G4
H1I3J2K3L9M2N1O1P1Q2R3S7T2U5V3W3X1Y1, 将15个标准品种的QR编码利用厦门联图网络科技有限公司开发的在线二维码生成软件(http://www.liantu.com/shiliang/)生成对应的矢量二维码, 并作图。
利用POPGEN 1.32软件对分析25个SSR标记在15个柱花草标DUS测试标准品种中的遗传多样性相关参数进行了分析(表2), 结果显示, 25个SSR标记在15个柱花草标准中均具有多态性, 共产生132个等位基因, 每个标记产生3~9个等位基因, 平均产生5.280个; 每个标记的有效等位基因数为1.751~7.258个, 平均3.498个; 每个标记的Nei’ s多样性指数为0.429~0.862, 平均0.680; 各SSR标记的Shannonx信息指数为0.820~2.084, 平均1.368; 另外, 各SSR标记的多态性信息含量(PIC)平均为0.638, 变化范围为0.393~0.847, 其中SSR58的PIC值最低, 为0.393, SSR35的PIC值最高, 为0.847, 25对SSR引物的多态性信息含量主要集中在0.600~0.800, 表明本研究中SSR标记表现出较高多态性。
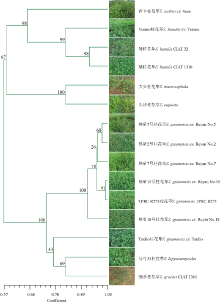
采用NTSYS-pc 2.10e软件中的非加权组平均法(UPGMA), 对15个柱花草DUS测试标准品种进行聚类分析(图1), 品种间的遗传相似系数介于0.500~0.992, 平均值为0.714, 聚类分析结果显示柱花草不同品种间的变异较大。当遗传相似系数为0.570时, 可将15个柱花草标准品种分为两个类群, 其中第1个类群主要包含西卡柱花草、有钩柱花草、矮柱花草、头状柱花草及大头柱花草, 而第2个类群主要为圭亚那柱花草, 另外还包括马弓形柱花草和细茎柱花草。在第1个类群中, 西卡柱花草、有钩柱花草及矮柱花草的遗传距离更为接近, 头状柱花草和大头柱花草同样能划分为1个亚群; 第2类群中, 圭亚那柱花草Tardio与马弓形柱花草和细茎柱花草可聚为同一亚类。
利用SSR标记对15份柱花草标准品种进行聚类分析的结果(图1)与热带牧草中心前期对192份柱花草种质资源聚类分析时的结果一致, 依照DUS测试指南中测试性状进行聚类分析后也可将不同种质资源划分为两个大的类群, 其中第1个类群以灌木型柱花草为主, 第2个类群主要是草本型柱花草[18]。性状测试与分子标记的聚类分析结果一致, 表明利用SSR标记构建指纹图谱作为柱花草品种评定标准具有可行性。
2.3 15份柱花草标准品种指纹图谱的构建
本研究中不仅运用25对核心SSR引物建立了15个柱花草DUS测试标准品种的指纹图谱(表3), 还依据DNA指纹图谱组成QR编码, 通过在线二维码生成软件对标准品种进行二维编码(图2), 最终构建成15份柱花草DUS测试标准品种DNA指纹图谱的QR二维码数据库。该数据库所包含的主要信息:编号为该标准品种在中国热带农业科学院热带作物资源品种研究所热带牧草研究中心柱花草种质资源的系统编号, 材料名称为标准品种名称, DNA指纹图谱为25对核心SSR引物及其对应的基因型编号, 如编号为2的柱花草标准品种, 其材料名称为西卡柱花草, 对应的DNA指纹图谱为A2B6C4D1E6F2G4H1I3J2K3L9M2N1O1P1Q2R3S7T2U5V3W3X1Y1。而DNA指纹图谱QR编码生成二维码在今后的育种工作中能够更方便更准确地通过扫描二维码获得该品种的相关信息, 这样为实现柱花草品种鉴定的自动化和信息化提供基础, 为今后品种鉴别和育种等工作提供直接证据。
| 表3 柱花草标准品种DNA指纹图谱 Table 3 The DNA fingerprinting of Stylosanthes sample cultivars |
随着作物种质资源的不断挖掘, 育种材料背景日益狭窄, 仅仅依靠目前传统的DUS测试技术以植物学性状进行品种鉴别的方法是不可取的。一方面, 植物学性状受环境影响较大, 影响DUS测试的准确性, 且观测周期长, 一般需要2~3年的田间重复试验; 另一方面, 标准品种存在不稳定的因素, 相近品种依据植物学性状分辨困难[3]。因此, 基于DNA多态性的分子标记技术正在成为植物新品种DUS测试的发展方向, RAPD、ISSR、AFLP和SRAP等分子标记已先后被用于植物品种鉴定[4]。这些标记均是采用无基因组序列信息的标记技术, 尽管有一定的实用性, 但标记随机性强、稳定差[17]。SSR技术具有共显性、重复性好等优点, 被《植物品种鉴定 DNA指纹方法 总则》NY/T2594-2014推荐为当前各植物品种DNA指纹鉴定的主要标记方法。
目前, SSR 标记在柱花草中主要应用于遗传多样性分析与核心种质的构建。Chandra等[17]利用BAC文库和EST序列开发了41个SSR标记, 并利用这些SSR标记对20份灌木柱花草种质进行了遗传多样性分析。Santos-Garcia等[31]利用20个SSR标记对150个圭亚那柱花草种质进行了遗传多样性分析, 将其分为9 类。Santos-Garcia等[32]利用SSR标记对134份大头柱花草种质和192份头状柱花草种质进行了遗传多样性分析, 最终确定了分别含有23份大头柱花草和13份头状柱花草的核心种质。
本研究用25个SSR标记对15份柱花草DUS测试标准品种进行检测, 共检测到132个等位基因, 每个标记可检测到3~9个等位基因, 平均为5.280个; 每个SSR标记的多态性信息含量为0.393~0.847, 平均为0.638, 说明SSR标记具有较高的遗传多态性。本研究中聚类分析表明, 25个SSR标记能有效地将15份柱花草DUS标准品种分为两大类, 该聚类结果与用其他分子标记分析的结果相一致[33, 34, 35]。
DNA指纹图谱的构建方法也有多种, 包括巫桂芬等[36]利用标记多态性产生的等位变异构建法, 刘娟等[37]运用Gel 2.0软件对特征引物扩增的凝胶带谱构建指纹模式图谱, 杨青松等[38]利用特征引物组合构建个体特异的DNA指纹图谱法以及本研究参考的范建光等[8]、宋海斌等[30]图谱代码构建法形成DNA指纹图谱的QR编码生成二维码等, 相比之下指纹图谱QR编码构建法包含信息量丰富, 准确可靠, 并且在实际生产中应用简便。
由于柱花草遗传学背景研究较少, 已报道的SSR标记数目有限, 本研究选取的25个SSR标记可能不能够很好的反映不同柱花草种质资源间的遗传差异, 今后应开发更多的SSR标记, 并将多态性高、条带清晰的SSR标记加入到已构建好的指纹图谱库中, 以更好地将SSR标记技术应用于柱花草的DUS测试和新品种鉴定。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|