第一作者:呼和(1976-),男(蒙古族),内蒙古通辽人,副研究员,博士,主要从事环境污染的微生物修复、农牧业固体废弃物循环利用和微生物生态研究。E-mail:[email protected]
使用荧光定量PCR法测定亚硝酸还原酶基因( nirK和 nirS)遗传特异性片段,研究了草原撂荒地土壤反硝化微生物丰度随着撂荒时间的变化。结果表明,轻度放牧草原和3种不同撂荒时间地的土壤中 nirK基因型反硝化微生物丰度都显著高于 nirS基因型反硝化微生物丰度( P<0.05)。土壤 nirK及 nirS基因型反硝化微生物丰度撂荒地和轻度放牧地之间均有显著差异( P<0.05),但3种撂荒地均未发生显著变化( P>0.05)。此外,这两种基因型反硝化微生物丰度之间有极显著线性负相关关系( P<0.001)。以上结果说明, nirK和 nirS基因型反硝化微生物对环境因子的响应有差异。
Determination of nitrite reductase gene ( nirK and nirS) specific fragment by real-time quantitative PCR method, we studied the dynamics of denitrification microbial abundance in abandoned land soil with different abandoned years. The results showed that the nirK genotype denitrifier abundance were significantly higher than nirS genotype denitrifier abundance in 3 kinds of abandoned land for 1, 5, and 25 years and light grazing grassland. Both of genotypes denitrifier abundance were significant difference between the light grazing grassland and the three abandoned land soils ( P<0.05), but there was no significant change among the 3 kinds of abandoned land soil. Moreover, there were extremely significantly negative linear relationship between the two genes abundance in the investigation soils ( P<0.001), indicating that the nirK and nirS show different response to the environmental factors on their growth.
由于人口迅速增长, 我国对粮食及经济作物的需求量日益增大, 因此, 需要多种途径扩大耕地面积, 如开垦荒地、围海造田、围湖造田等, 这就造成了草地退化等现象, 如开垦已成为呼伦贝尔草原沙化、退化的主要原因之一[1]。近年来, 国家开始实施退耕还林、还草等工程, 以改善生态环境[2]。目前, 国内外关于退耕地的研究主要集中于植被群落的演替[3]、土壤理化性质的变化[4]以及种植植物的影响[5, 6]等方面, 但随着退耕时间的延长, 土壤微生物群落如何变化, 尤其是关于微生物氮循环重要环节之一— — 反硝化基因丰度变化及其相互关系的研究鲜见报道。生物反硝化作用是氮循环的主要环节之一, 反硝化作用在土壤中普遍存在, 其主要是通过微生物逐步地还原氮氧化合物, 释放出一氧化氮(NO)、氧化亚氮(N2O)和氮(N2)等, 从而导致土壤中氮元素的减少, 而且, N2O和NO的释放可能导致全球变暖[7]。反硝化微生物广泛分布于细菌和古菌中, 在几种真菌的线粒体中也发现了反硝化作用[8]。反硝化作用主要指在硝酸还原酶(Nar)、亚硝酸还原酶(Nir)、氧化亚氮还原酶(Nor)和一氧化二氮还原酶(Nos)等的作用下将硝酸盐(N
研究区域位于内蒙古自治区东北部的呼伦贝尔草原新巴尔虎右旗境内(116° 40' 18''-117° 01' 56'' E, 48° 32' 00''-48° 38' 50'' N), 为半干旱草原。试验共设4个处理, 3个撂荒农田和1个轻度放牧草地。3个撂荒农田的撂荒时间分别为1、5和25年(Y1、Y5和Y25), 轻度放牧草地(LG)设为对照, 放牧率为1.4只羊· hm-2。在Y1、Y5和Y25样点, 玉米(Zea mays)、向日葵(Helianthus annuus)、圆柱披碱草(Elymus cylindricus)无规律轮作约40年, 各样地之间距离< 10 km, 且各样点地势相似, 均为平地。各样地2000-2009年平均温度和降水量分别为1.6 ℃和213 mm, 海拔在545~568 m, 均为沙质土壤。
在每个样点随机设5个1 m× 1 m的样方, 于2010年8月进行植被调查和土壤取样。记录每个样方内的植物物种组成, 测定样方的盖度、高度和密度, 并计算Shannon-Wiener多样性指数。剪取植物地上部分, 按物种分类, 60 ℃下干燥48 h, 称重, 测定地上植物干重。土壤湿度用TRIME-FM (Ettlingen, Germany)测定。另外, 在每个样方随机选择5个点收集0-10 cm土样, 混合为一个样品, 并移去表面有机质和细根。把每个混合样品分为两部分, 一部分过2 mm筛后密封于15 mL离心管中, 放在液氮罐里运回实验室, 储存于-80 ℃冰箱中待用于微生物试验, 另一部储存于4 ℃下, 用于测定土壤理化特性。土壤KCl浸提物中的NO3-N和NH4-N分别用锌还原-萘乙二胺法[15]和靛酚蓝比色法[16]测定, 土壤磷含量用Truog法测定[17], 有机碳和总氮使用NC分析仪(Sumigraph NC-900; Sumika Chemical Analysis Service, Tokyo, Japan)采用干燃烧法测定[18]。土壤pH采用电位法测定。
每个土样分别称取3份, 每份0.5 g, 采用FastDNA Spin Kit(MP Biomedicals, Illkirch, France)提取土壤DNA, 实验步骤按照说明书进行。采用SmartSpec Plus spectrophotometer (Bio-Rad Laboratories, United States)测定DNA的纯度及浓度。样品保存于-20 ℃冰箱中备用。
采用实时定量PCR(Applied Biosystems StepOne Plus 96-well, 美国)与预混液(Applied Biosystems Power SYBR Green, 美国)相对定量nirK和nirS基因片段, 各反应的引物、qPCR反应条件、扩增片段大小、标准微生物及混合体系组成如表1和表2所示。qPCR反应开始前先在95 ℃下进行10 min的酶活化。扩增产物的特异性通过观察熔解曲线上的单一溶解峰和溴化乙锭染色的2%琼脂糖凝胶上特异条带的出现来检测。连续稀释(10~107拷贝数· μ L-1)标准细菌对应基因PCR扩增产物制作各基因的标准曲线, 使用Eff=[10(-1/slope)-1]公式计算nirK和nirS基因扩增效率(Eff), 分别为92.0%和98.2%。每个反应3个重复, 平均拷贝数转换为每克干土样中的基因拷贝数。
| 表1 nirK和nirS基因的qPCR条件及引物 Table 1 qPCR Condition of nirK and nirS gene and primer |
| 表2 nirK和nirS基因的qPCR反应体系 Table 2 qPCR reaction system of nirK and nirS gene |
qPCR数据和环境因子特征采用SPSS 19.0(SPSS, Inc., Chicago, IL, 美国)进行方差分析(ANOVA)。ANOVA分析采用了Tukey’ s test检验。如果每组数据不是正态分布, 在分析前将数据进行对数变换。采用SIMCA-P 11.5(Umetrics AB, Umea, 瑞典)分析软件, 偏最小二乘法(PLS)分析nirS和nirK基因拷贝数与环境因子之间的回归系数和变量投影重要性(VIP)。
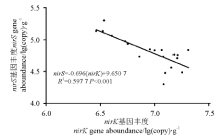
开垦耕种活动显著影响了nirK和nirS基因丰度比例(图1)。撂荒地nirK基因的丰度是nirS基因的1 000倍左右, 然而, 在LG土壤中, nirK基因丰度是nirS基因10倍左右。随着撂荒时间的延长, 这两种基因丰度都没有显著变化(P> 0.05), 但是, 3个撂荒地的nirK基因的丰度均显著高于LG的(P< 0.05)。另外, 4个样地亚硝酸还原酶基因nirK的拷贝数和nirS基因的拷贝数存在极显著线性关系(图2), 回归方程为nirS=-0.696 (nirK)+9.650 7(R2=0.597 7, P< 0.001)。
两种亚硝酸还原酶基因(nirK和nirS)都与NH4-N、有机碳、总氮、植物多样性和地上生物量显著相关, VIP值均大于1(图3)。由回归系数值可知, nirK基因拷贝数与NH4-N、有机碳、总氮和地上生物量呈负相关, 与植物多样性呈正相关, 而nirS基因的拷贝数与nirK基因的相关性分析结果正好相反。此外, pH、NO3-N、有效磷和H2O等环境因子对nirK和nirS基因的拷贝数无显著影响, VIP值都小于1。
土壤氮循环主要依赖于微生物的氮循环作用, 即固氮、亚硝化、硝化及反硝化等作用把不同类型的氮素化合物改变为亚硝酸根、硝酸根、NO及N2O的形式。其中, 具有亚硝酸还原酶基因的微生物能把亚硝酸盐还原为NO, 是生物氮循环这一过程的重要影响环节之一, 与其它参与生物氮循环基因相同, 亚硝酸还原酶基因也是研究者关注的重要反硝化功能基因。因此, 本研究以nirK和nirS基因作为研究对象, 采用实时荧光PCR法探讨了不同类型亚硝酸还原酶基因丰度在呼伦贝尔草原不同撂荒时间耕地土壤中的动态变化以及其与环境因子之间的关系。
研究发现, 撂荒地和轻度放牧草原的土壤理化特征及植被状况差异较大[19]。3个撂荒地的地上生物量有显著差异(P< 0.05), 但铵态氮、有效磷、有机碳和植物多样性无显著差异(P> 0.05)[19]。此外, Y5样地的土壤pH和硝酸盐氮(NO3-N)显著高于Y1和Y25, Y1和Y25之间无显著差异。Y5的总氮显著高于Y1的(P< 0.05), 但Y25的与Y1和Y5的均无显著差异。Y25的土壤水分含量(H2O)显著高于Y1的, Y5的与Y1、Y25的均无显著差异[19]。
在3个不同撂荒时间的撂荒地土壤中, nirK基因丰度显著高于nirS基因, 前者均约为后者的103倍, 而在轻度放牧草原土壤中, 前者约为后者的10倍, 这说明该研究地土壤中具有nirK基因的微生物是主要的亚硝酸还原微生物。在森林土壤(壤土和粉壤土)[20]和稻田土壤[21]中也得到了相同的结果, 即nirK基因丰度高于nirS基因丰度。此外, 在撂荒地土壤中nirK和nirS基因丰度分别显著高于和低于轻度放牧草原。根据PLS分析的结果, 植物多样性和nirK基因丰度之间有显著正相关(VIP=1.05), 表明nirK类型反硝化微生物有可能很容易受不同组成和数量的根系分泌物的影响, 在沙壤土中, 随着植被多样性的变化其土壤微生物群落结构也变化[22], Bremer等[23]使用狐尾草(Alopecurus pratensis)、黄花茅(Anthoxanthum odoratum)、燕麦草(Arrhenatherum elatius)、绒毛草(Holcus lanatus)、老鹳草(Geranium pratense)、车前(Plantago lanceolata)、毛茛(Ranunculus acris)和蒲公英(Taraxacum officinale)的微宇宙试验中发现, 不同植物微宇宙土壤有不同的nirK群落结构。Baneras等[24]也指出植被类型的改变对nirK类型反硝化微生物群落具有显著影响。除此之外, NH4-N、有机碳、总氮和地上生物量对于nirK和nirS基因丰度分别呈显著负相关和正相关关系(VIP > 1)(图3), 说明亚硝酸还原细菌的丰度变化受到土壤不同理化性质的共同影响, 导致两种类型亚硝酸还原细菌丰度呈此消彼长的趋势, nirK和nirS基因丰度之间呈极显著线性关系(P< 0.001)(图2)。许多研究[25, 26, 27]表明, 土壤微生物的分布、活动、多样性以及丰度与土壤理化性质密切相关。Maag和Vinther[28]在森林土壤中发现, 反硝化速率受多个环境因子联合调控。 尽管nirK和nirS基因与NO3-N的含量密切相关, 而本研究未发现nirK和nirS基因丰度与NO3-N含量具有显著相关性, 这有可能是在自然环境土壤中不同环境因子共同影响亚硝酸还原微生物的结果。许多研究者也在不同环境的土壤中发现了反硝化细菌群落与NO3-N含量具有很弱的相关性[29, 30, 31]。此外, Kong等[32]认为, 在亚硝酸还原微生物生存的微生境中, 其变化有可能与NO3-N含量变化无关。
总之, 在本研究区的撂荒地, nirK和nirS基因型反硝化微生物丰度随着撂荒时间无有显著变化, 然而它们之间有显著线性关系, 两种基因型反硝化微生物随着环境因子的变化, 有可能互补调节亚硝酸盐还原为NO的作用。但本研究只分析了DNA水平上的反硝化微生物丰度, 其活性及表达的规律还有待RNA样品来分析说明。另外, 尚需开展nirK和nirS基因型反硝化微生物群落结构和分布, 以便了解随着撂荒时间的延长反硝化微生物群落的演替规律, 更科学地通过调节反硝化作用管理撂荒地。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|