刘文献(1981-),男,河南开封人,副教授,博士,主要从事牧草育种与分子生物学研究。E-mail:[email protected]
第一作者:齐晓(1982-),男,河北泊头人,农艺师,在读博士生,主要从事草品种管理和推广工作。E-mail:[email protected]
共同第一作者:张正社(1990-),男,甘肃靖远人,在读博士生,主要从事牧草育种及牧草种子分子生物学研究。E-mail:[email protected]
碱性亮氨酸拉链(bZIP)转录因子是真核生物转录因子中分布最广泛、最保守的一类蛋白。目前在许多植物中已发现大量的bZIP转录因子,这些bZIP转录因子成员广泛参与种子贮藏基因的表达、植物的生长发育、光信号传导、病害防御、生物和非生物胁迫应答以及ABA的敏感性等各种信号的反应。本研究首次从紫花苜蓿( Medicago sativa)全转录组水平鉴定出bZIP转录因子家族共包含138个基因,根据bZIP蛋白序列进行系统进化分析可以将其分为10类;对 MsbZIP基因的系统进化分析表明该基因家族在分类上有很高的保守性。该转录因子家族的基因密码子偏好性分析表明, MsbZIP基因密码子偏好使用A/T碱基。此外, MsbZIP基因GO功能注释分析结果显示,138个 MsbZIP基因最终分为23个GO分类,总体包括分子功能和生物学过程两类。相关性分析结果表明,共有372对基因表达相关性极显著( P<0.01)。本研究可为紫花苜蓿bZIP转录因子功能特性、进化历程和生物功能的深入研究奠定基础。
Basic leucine zipper transcription factors are one of the most extensive and conserved transcription factors in the eukaryote proteins. Currently, several bZIP transcription factors have been identified in many plants. bZIP transcription factors participate extensively in the regulation of seed storage gene expression, development, photomorphogenesis, pathogen defense, biotic and abiotic stress responses, ABA signalling, and more. In this study, 138 bZIP transcription factor-encoding genes were identified by a comprehensive computational analysis of the Medicago sativa transcriptome databases. Phylogenetic analysis of the bZIP protein sequences permits classification of the genes into 10 groups in M. sativa. The codon bias results showed that the alfalfa MsbZIP gene was biased toward the synonymous codons with A and T. Moreover, the gene ontology (GO) term annotations of the 138 MsbZIP were annotated and classified into 23 categories according to biological processes and molecular function. The co-expression relationship between MsbZIP genes showed that significant correlations existed between 372 pairs of MsbZIP genes at the 0.01 level. These results will be important for the functional characterization of bZIP transcription factors in M. sativa, and facilitate further research on the bZIP gene family regarding their evolutionary history and biological functions.
转录因子对植物众多重要的生物学过程起关键性调控作用[1, 2]。研究转录因子的转录调控网络及其生物学过程是了解转录因子功能特性的本质。截至目前, 植物中至少有64个转录因子家族被鉴定[3]。作为植物中最大的转录因子家族之一, 碱式亮氨酸拉链有多个不同的分类[4]。bZIP蛋白具有一个4060个氨基酸组成的保守结构域, 其包含1个碱性DNA结合域, 可通过一个固定的N-x7-R/K结构与特异DNA序列结合; 还包含一个亮氨酸拉链二聚体结构域与碱性区紧密结合, 每7个氨基酸的第7位有一个亮氨酸以及其它疏水性残基位第3和第4位, 亮氨酸拉链形成一个两亲性的α 螺旋, 可影响bZIP蛋白与DNA结合之前的二聚化[5]。该转录因子家族除了bZIP结构域外还包含其它具有转录活化功能的结构域, 例如, R/KxxS/T和S/TxxD/E结构域, 分别是Ca+独立蛋白激酶和酪蛋白激酶Ⅱ 的磷酸化位点[6]。此外, 脯氨酸富集区、谷氨酰胺富集区以及酸性结构域在bZIP基因的转录活化过程中也具有重要作用[7]。
截至目前, bZIP基因家族在多种植物全基因组水平得到了综合鉴定和预测。例如, 在拟南芥(Arabidopsis thalianna)中鉴定出75个基因[5], 葡萄(Vitis vinifera)中55个[8], 水稻(Oryza sativa)中89个[9], 大豆(Glycine max)中131个[7], 高粱(Sorghum vulgare)中92个[10], 玉米(Zea mays)中125个[11], 蒺藜苜蓿(Medicago truncatula)中75个[12]。研究证明, bZIP可以通过与启动子区域的顺式作用元件相互作用, 抑制或激活多个下游基因表达, 从而参与转录调控过程。bZIP转录因子参与多个生物学过程, 包括种子萌发与成熟、植物衰老、光形态建成、糖代谢等, 特别是在非生物胁迫的抗逆反应中起重要作用, 例如盐胁迫、干旱胁迫、低温胁迫、热激反应和生物胁迫等[13, 14, 15, 16]。
豆科植物是第三大开花植物, 包含了大约650属18 000种。就经济价值而言, 豆科作物仅次于禾本科作物, 排行第二, 占世界作物总产值的27%[17]。紫花苜蓿(M. sativa)是全球栽培面积最大的牧草, 大约有3 000万h
112 262条紫花苜蓿unigene序列获取自AGED数据库(http://plantgrn.noble.org/AGED/)[20]。利用已鉴定出的75条蒺藜苜蓿bZIP转录因子cDNA序列同紫花苜蓿unigene序列进行BLAST(E-value=1e-005), 搜索潜在的包含bZIP转录因子的紫花苜蓿unigene[12]。利用NCBI-CDD在线工具(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)对搜索出的unigene进行保守蛋白结构域鉴定。利用ORF Finder在线工具(http://www.ncbi.nlm.nih.gov/orffinder/)从unigene中获取CDS序列。经过去冗余后, 最终共鉴定出138个假定的MsbZIP转录因子基因。
利用MUSCLE在线工具(http://www.ebi.ac.uk/Tools/msa/muscle/)进行多序列比对, 系统进化树利用MEGA 7.0(http://www.megasoftware.net/)邻接法构建, 并对构建的进化树进行自举评估(Boot-strap), 迭代次数为1 000, 其它参数使用系统默认值。利用蒺藜苜蓿和紫花苜蓿的bZIP蛋白序列进行聚类分析。MsbZIP蛋白结构域通过MEME在线工具(http://meme-suite.org/tools/meme)查询。
利用CodonW软件计算有效密码子数(Enc)、CDS区的GC含量、密码子中第3位碱基的GC含量(GC3s)和相对同义密码子使用度RSCU(Relative synonymous codon usage)。
紫花苜蓿基因表达谱数据下载自AGED数据库(http://plantgrn.noble.org/AGED/index.jsp)。利用WEGO在线工具(http://wego.genomics.org.cn/cgi-bin/wego/index.pl)进行GO分类。MsbZIP基因组织表达相关性利用R语言包分析作图。
bZIP保守结构域有两个特征结构(碱性区域和亮氨酸拉链), 这在拟南芥[5]、水稻[9]、葡萄[8]、高粱[10]、玉米[11]和蒺藜苜蓿[12]中都已得到系统的分析。本研究通过构建本地BLAST数据库, 与蒺藜苜蓿bZIP蛋白序列进行tblastn比对, 共鉴定出138个非冗余bZIP基因。通常bZIP基因按照每个基因在染色体上的位置顺序命名[5], 但由于紫花苜蓿缺乏染色体数据, 因此利用blast得到的contig先后顺序将紫花苜蓿bZIP基因命名为MsbZIP1-MsbZIP138(表1)。利用在线工具ExPASy对紫花苜蓿bZIP蛋白质大小、分子量、理论等电点及平均亲水性等理化性质进行分析发现, 最长的蒺藜苜蓿bZIP蛋白(MsbZIP93)包含766个氨基酸残基, 最短的蒺藜苜蓿bZIP蛋白(MsbZIP138)仅有76个氨基酸残基; 分子量在8 789.3(MsbZIP138)84 011.8 D(MsbZIP93); 其理论等电点范围为4.67(MsbZIP126)11.34(MsbZIP44); 所有bZIP蛋白均表现为亲水性(表1)。
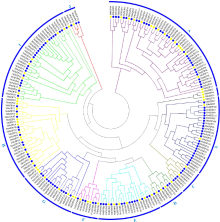
为了研究bZIP转录因子在紫花苜蓿和蒺藜苜蓿中的系统进化关系, 利用紫花苜蓿中的138条蛋白序列和蒺藜苜蓿中的75条蛋白序列用于系统进化树的构建。基于分析, 将进化树分为10个分支(A-J)(图1)。在这10个分类中, A类中所包含的紫花苜蓿bZIP基因数目最多, 有31个; J类仅包含3个紫花苜蓿bZIP基因, 在所有分类中最少。本研究的分类结果同以前在蒺藜苜蓿中的研究有所不同[12]。例如, MsbZIP56、MsbZIP60、MsbZIP68和MsbZIP20、MsbZIP39、MsbZIP48、MsbZIP52在以前的研究中都在同一分类, 本研究中被分到A类和I类。
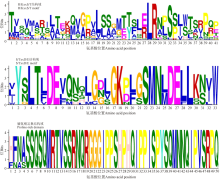
bZIP结构域是bZIP转录因子家族的核心结构域, 它可以优先地结合特异性顺式作用元件来调控基因的表达。然而, bZIP基因家族的功能多样性也由bZIP蛋白的其它保守结构域所影响[21]。本研究运用MEME在线分析工具对MsbZIP转录因子家族中其它保守结构域进行了搜索。如图2所示, 结构域a和b拥有R/KxxS/T和S/TxxD/E位点, 已有研究表明, 这两个位点分别是Ca2+独立蛋白激酶和酪蛋白激酶Ⅱ 的磷酸化关键位点[6, 22]。拥有这两种结构域的MsbZIP蛋白分别有42和10个。结构域c中富含脯氨酸, 是个典型的脯氨酸富集结构域。类似的结构域在拟南芥bZIP转录因子中已被鉴定, 并且研究表明其具有转录激活的潜能[23]。
ENc值(effective number of codons)为基因的密码子偏好性程度提供了一个客观的评判标准, 代表特定基因中同义密码子非均衡使用的偏好程度。该值在20(每个氨基酸只使用1个密码子的极端情况)到61(各个密码子均被平均使用)之间, 越靠近20偏好性越强。GC3s则表示密码子的第3位碱基中G+C的含量在第3位碱基总量中所占的比率[24]。本研究应用CodonW程序计算紫花苜蓿bZIP基因的ENc值、GC含量和GC3s值分别为50.71、0.438和0.340, ENc值大于50, 表明紫花苜蓿bZIP基因各密码子在编码氨基酸时出现的频率比较一致; 紫花苜蓿bZIP基因编码区GC含量较低, 而GC3s值则更低, 表明紫花苜蓿偏好使用以A、T结尾的密码子, 且在整个编码区序列中A+T含量大于G+C。
| 表1 紫花苜蓿bZIP转录因子家族信息 Table 1 Information of bZIP transcription factor family in Medicago sativa |
2.5 紫花苜蓿基因相对同义密码子使用度分析 相对同义密码子使用度(relative synonymous codon usage, RSCU)是指对于某一特定的密码子在编码对应氨基酸的同义密码子中的相对概率。RSCU值与氨基酸的使用及密码子的丰度无关, 它能直观地反映出密码子使用的偏好性程度[25]。如果密码子的使用没有偏好, 则该密码子的RSCU=1。当某一密码子的RSCU值大于1, 则表明该密码子的使用频率相对较高, 反之亦然。CodonW程序计算结果(表2)表明, 在紫花苜蓿bZIP基因的密码子中, 有偏好性(RSCU> 1)的密码子共计27个, 其中以G/C结尾的2个, 以A/T结尾的25个。各氨基酸RSCU值最高的密码子分别是:TTT(Phe)、TTG(Leu)、ATT(Ile)、GTT(Val)、TCA(Ser)、CCT(Pro)、ACT(Thr)、GCT(Ala)、TAT(Tyr)、CAT(His)、CAA(Gln)、AAT(Asn)、AAA(Lys)、GAT(Asp)、GAA(Glu)、TGT(Cys)、AGA(Arg)、GGT/GGA(Gly)、TGA(终止密码子)。这20个密码子以G/C结尾的1个, 以A/T结尾的19个。以上结果同样表明, 紫花苜蓿偏好于以A/T结尾的密码子。
2.5 紫花苜蓿基因相对同义密码子使用度分析 相对同义密码子使用度(relative synonymous codon usage, RSCU)是指对于某一特定的密码子在编码对应氨基酸的同义密码子中的相对概率。RSCU值与氨基酸的使用及密码子的丰度无关, 它能直观地反映出密码子使用的偏好性程度[25]。如果密码子的使用没有偏好, 则该密码子的RSCU=1。当某一密码子的RSCU值大于1, 则表明该密码子的使用频率相对较高, 反之亦然。CodonW程序计算结果(表2)表明, 在紫花苜蓿bZIP基因的密码子中, 有偏好性(RSCU> 1)的密码子共计27个, 其中以G/C结尾的2个, 以A/T结尾的25个。各氨基酸RSCU值最高的密码子分别是:TTT(Phe)、TTG(Leu)、ATT(Ile)、GTT(Val)、TCA(Ser)、CCT(Pro)、ACT(Thr)、GCT(Ala)、TAT(Tyr)、CAT(His)、CAA(Gln)、AAT(Asn)、AAA(Lys)、GAT(Asp)、GAA(Glu)、TGT(Cys)、AGA(Arg)、GGT/GGA(Gly)、TGA(终止密码子)。这20个密码子以G/C结尾的1个, 以A/T结尾的19个。以上结果同样表明, 紫花苜蓿偏好于以A/T结尾的密码子。 紫花苜蓿bZIP基因的密码子用法 Table 2 Codon usage of Medicago sativa coding sequences |
为了预测MsbZIP基因潜在的功能, 利用MsbZIP基因的GO数据通过WEGO在线程序对紫花苜蓿bZIP基因进行功能注释(图3)。MsbZIP基因总体包括分子功能和生物学过程两类。138个MsbZIP基因最终分为23个GO分类。在分子功能分类中, 具有分子结合功能的基因最多, 有127个, 占92%。其次是具有转录调节活性的基因, 有119个, 占86.2%。仅有1个基因具有分子结构功能, 所占比例最少。基于生物过程, 这些基因共分为18类。其中有129个基因具有代谢过程和细胞过程功能, 所占比例最高, 为93.5%; 具有生长功能的基因仅有1个, 所占比例最少, 为0.7%。
为了研究MsbZIP基因间的共表达关系, 基于MsbZIP基因的转录组数据计算了皮尔逊相关系数(PCC)。结果表明, 共有372对基因表达相关性极显著(P< 0.01, 双尾检验), 其中有355对(95.43%)基因表达极显著正相关(r> 0.9), 17对(4.57%)基因表达极显著负相关(r< -0.9)。这表明这些基因可能参与同一生物过程的调控。进一步对40对同源基因(MsbZIP56/MsbZIP104, MsbZIP106/MsbZIP113, MsbZIP25/MsbZIP89, MsbZIP16/MsbZIP19, MsbZIP117/MsbZIP11, MsbZIP126/MsbZIP41, MsbZIP28/MsbZIP108, MsbZIP42/MsbZIP52, MsbZIP32/MsbZIP61, MsbZIP75/MsbZIP114, MsbZIP35/MsbZIP85, MsbZIP118/MsbZIP116, MsbZIP14/MsbZIP22, MsbZIP76/MsbZIP112, MsbZIP48/MsbZIP08, MsbZIP09/MsbZIP129, MsbZIP10/MsbZIP34, MsbZIP49/MsbZI-P97, MsbZIP71/MsbZIP79, MsbZIP62/MsbZIP103, MsbZIP83/MsbZIP115, MsbZIP36/MsbZIP46, MsbZIP107/MsbZIP123, MsbZIP68/MsbZIP98, MsbZIP73/MsbZIP81, MsbZIP04/MsbZIP23, MsbZIP50/MsbZIP47, MsbZIP72/MsbZIP110, MsbZIP40/MsbZIP119, MsbZIP63/MsbZIP66, MsbZIP55/MsbZIP93, MsbZIP31/MsbZIP77, MsbZIP26/MsbZIP54, MsbZIP58/MsbZIP92, MsbZIP06/MsbZIP86, MsbZIP88/MsbZIP124, MsbZIP34/MsbZIP18, MsbZIP64/MsbZIP65, MsbZIP100/MsbZIP121, MsbZIP59/MsbZIP105)进行了相关性分析, 如图4所示。从对角线的方块区域可以看出这40个同源基因对可以分为6组。除了旁系同源基因对MsbZIP56/104, MsbZIP49/97, MsbZIP36/46, MsbZIP63/66, MsbZIP31/77, MsbZIP26/54, MsbZIP58/92和MsbZIP134/18分布在不同的组, 其它旁系同源基因对均在同一组中(图4)。相关性分析表明, 分布在同一组的旁系同源基因对同样也具有很高的相关性。例如, MsbZIP106与MsbZIP113(r=0.991, P< 0.01)正相关, MsbZIP25与MsbZIP89(r=0.979, P< 0.01)正相关。相反, 非旁系同源基因之间也有相关性很高的情况。例如, MsbZIP88与MsbZIP16(r=-0.898, P< 0.05)负相关, MsbZIP92与MsbZIP19(r=-0.831, P< 0.05)负相关, MsbZIP28与MsbZIP41(r=0.815, P< 0.05)负相关。
bZIP蛋白家族广泛存在于真核生物中, 响应多种生物和非生物胁迫反应, 能调控生物的生长发育过程。bZIP基因在植物中分布广泛, 通过多种植物研究发现, 该类蛋白种类及数量在物种间存在差异[26]。随着大量紫花苜蓿转录组测序结果的公布, 利用生物信息学技术可对紫花苜蓿bZIP蛋白家族进行全面系统地分析[27, 28, 29]。
本研究首次通过生物信息学手段对紫花苜蓿的转录组数据进行比对分析, 从中鉴定出138个紫花苜蓿bZIP基因, 比蒺藜苜蓿所鉴定的bZIP基因(75个)多一倍, 这可能是紫花苜蓿基因组发生加倍所造成的。通过聚类分析与蒺藜苜蓿bZIP基因家族相比较, 可将紫花苜蓿bZIP基因分为10个亚类。在这些亚类中, A和I类所拥有基因数目最多, 分别有31个和25个基因, J组仅有3个基因, 分别是MsbZIP55、MsbZIP93和MsbZIP128。在所有分类中, 都包含了蒺藜苜蓿基因, 说明紫花苜蓿和蒺藜苜蓿具有较高的同源性。同源性较高的基因, 其功能也可能具有相似性。例如MsbZIP61与MsbZIP137具有较高同源性, 而MsbZIP61基因与盐胁迫相关, 推测MsbZIP137基因也可能与盐胁迫相关。MsbZIP23与MsbZIP38同源性较高, 而MsbZIP47与MsbZIP132同源性较高, MsbZIP23和MsbZIP47基因受干旱胁迫诱导, 推测MsbZIP38和MsbZIP132也可能与干旱胁迫相关[12]。
通过motif比对分析发现, 在bZIP蛋白家族中除了主要结构域外还含有其它一些结构域。这些结构域中, 最典型的R/KxxS/T和S/TxxD/E与磷酸化相关。本研究中发现紫花苜蓿有42个bZIP蛋白包含R/KxxS/T, 有10个蛋白包含S/TxxD/E。结合前人研究结果可知, ABA的调控与磷酸化密切相关, 从而推测具有这些结构域的基因可能与植物的抗逆性有关。在蒺藜苜蓿bZIP基因家族的研究中也发现类似结果[12]。 密码子偏好性在蛋白质的结构功能、生物信息学等研究中具有重要意义, 受到了广泛的关注。研究发现, 密码子使用偏好性受多种因素影响, 例如基因碱基组成、表达水平等[30]。前人研究表明, 禾本科植物少用或避免使用以A或T结尾的密码子, 偏好于使用以G或C结尾的密码子, 而双子叶植物则相反[31, 32]。这与紫花苜蓿bZIP基因的密码子偏好性结果相一致(表2)。
GO功能分类是在某一功能层次上统计蛋白或基因数目及其组成的方法。通过GO分类发现, 共有138个MsbZIP基因与23个GO分类匹配。其中生物学过程的GO分类数多于分子功能的, 共有18个分类。说明紫花苜蓿bZIP转录因子在参与多个生物学过程调控中起着较为重要的作用。
表达具有正相关的基因在生物学过程中相互之间可能具有相互促进作用, 而负相关则预示着基因间可能相互抑制[33]。MsbZIP基因的转录组数据相关性分析表明, 这些相关性很高的基因可能共同参与调控相关的生物过程, 类似结果在葡萄bZIP转录因子中也曾被报道[8]。紫花苜蓿40对同源基因组织相关性结果表明, 大多数同源基因的功能具有相似性, 可能主要是因为这些旁系同源基因对之间具有很高的相关性, 但有少数同源基因的功能也表现出不同。
本研究首次在全转录组水平对MsbZIP基因的系统进化、蛋白保守结构域、密码子偏好性及基因表达等方面进行了系统分析, 这也是首次对紫花苜蓿基因家族进行系统分析。研究结果显示, 该转录因子家族可被划分为10类, 序列相对保守的基因分到同一类。蛋白保守结构域分析表明, 特定的结构域具有特定的功能。密码子偏好性分析表明MsbZIP基因密码子更偏向于使用A/T碱基。基因的转录组数据分析表明, MsbZIP基因在功能上具有较高的相关性。本研究结果可对紫花苜蓿bZIP基因家族基因功能的深入研究奠定一定基础。
(责任编辑 王芳)
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|