第一作者:许利英(1991-),女,四川南部人,在读硕士生,主要从事牧草种质资源研究。E-mail:[email protected]
采用EST-SSR分子标记对来自四川、西藏、青海的24份中华羊茅( Festuca sinensis)进行了遗传多样性和亲缘关系的研究。研究结果表明,筛选的17条引物共检测到102个位点,多态性位点每条引物5~8个;平均多态性位点6个,平均多态性位点百分率(PPB)93%;遗传距离变幅范围0.188~0.675,平均值0.486,多态性信息含量指数PIC值在0.240~0.470变化,平均值为0.381,这揭示了供试的中华羊茅种质资源具有较为丰富的遗传多样性。聚类分析和主成分分析结果表明,供试材料间亲缘关系与其地理来源等相关性较低,其中I-2-23-7和I-14-1-1野生材料表现出了独特的遗传特性,与其他供试材料间均具有较远的亲缘关系。通过地理海拔的分析,发现分布于中度海拔(3 000-3 500 m)的中华羊茅种质具有最高的Nei’s基因多样性(0.304 3)和香农多样性(0.462 2),遗传变异较高。本研究结果为青藏高原中华羊茅种质资源的保护和利用提供了重要的参考。
In this study, EST-SSR markers were used to study the genetic diversity and genetic relationship of 24 Festuca sinensis germplasms from Sichuan, Tibet and Qinghai. There were 102 loci detected from 17 primers and 5~8 loci per polymorphic locus. The mean number of polymorphic loci was 6 and the mean percentage of polymorphic loci was 93%. The range of genetic similarities was 0.188~0.675, with an average of 0.486, and polymorphism information content was 0.381, ranging from 0.240 to 0.470, which also revealed that the genetic resources of F. sinensis were rich in genetic diversity. The results of cluster analysis and principal component analysis showed that the association between the genetic relationships and geographical origin was low. Among them, the I-2-23-7and I-14-1-1 showed genetic specificity. The test materials were highly dissimilar between the 24 F. sinensis germplasms. Analysis of geographical elevations revealed that the F. sinensis germplasms with medium altitude (3 000-3 500 m) displayed the highest Nei’s gene diversity (0.304 3) and Shannon’s diversity index of 0.462 2, which indicated high genetic variation. The results provide an important reference for the conservation and utilization of the F. sinensis germplasm resources in the Qinghai-Tibet Plateau.
中华羊茅(Festuca sinensis)是禾本科羊茅属多年生草本植物, 是中国西北、华北、东北和青藏高原等地常见的野生牧草, 常生于海拔2 600~4 800 m的高山草甸、山坡草地、灌丛、林下等地。中华羊茅作为高寒牧区草地生产建设的优良牧草, 具有营养价值高、适口性好、再生性和抗逆性强等特点。茎秆柔软, 马、牛、羊都比较喜食, 其中在夏、秋、春三季是各类牲畜的主要牧草, 易增膘长肉。同时, 中华羊茅适应性广, 耐寒性和刈割能力强, 是高寒牧区草地建设、退牧还草和生态治理工程中最适宜的优良牧草品种之一, 也是我国青藏高原地区代表性的优良牧草之一[1, 2, 3]。因此, 开展中华羊茅种质资源及其适应性的研究对我国高寒牧区畜牧业的发展以及生态恢复建设具有重要意义
目前对中华羊茅的研究多集中在栽培草地建设、抗逆性及内生真菌等方面[4, 5, 6, 7], 但关于其种质资源遗传多样性方面的研究还未见报道。王康英[4]发现, 在3种钠盐(NaCl、NaHCO3、Na2CO3)胁迫下中华羊茅种子的发芽率、发芽势、发芽指数和活力指数均随着盐浓度的升高而降低。汪建军等[5]对温度和PEG处理对中华羊茅种子萌发的影响进行系统的分析, 表明低浓度(-0.2 MPa)的PEG溶液对中华羊茅种子活力具有促进作用, 高浓度(-0.8 MPa)的PEG溶液则对其种子活力有抑制作用。
目前分子标记技术已被广泛用于羊茅属物种的遗传育种研究, 特别是在高羊茅等代表性物种上的相关研究较多[8, 9, 10, 11]。其中SSR标记应用最为广泛, 其具有重复性好, 结果可靠性高, 操作简单; 多态性丰富且标记数量多等特点。在植物遗传育种研究中作为一种主要的分子标记技术已广泛应用于遗传图谱的构建、遗传多样性分析和系统学研究[12]。本研究采用EST-SSR分子标记技术对来自青藏高原地区的24个中华羊茅种质资源进行遗传多样性的评价分析, 旨在为中华羊茅野生种质的鉴定、保护及利用提供理论依据。
24份中华羊茅样品分别采集自四川、西藏和青海等青藏高原地区(表1), 其中23份为野生种质资源, 1份为登记品种。供试材料种子于2016年12月播种于花盆中, 放置在人工气候培养箱内, 待其生长到3~4片叶时取样。
| 表1 中华羊茅材料信息 Table 1 Data concerning Festuca sinensis |
每份中华羊茅材料选取10~15个单株的叶片混合, 采用植物基因组DNA提取试剂盒(DP305, 由天根生化科技有限公司提供)提取样品的DNA, 采用1%的琼脂糖凝胶电泳和超微量分光光度计(NANODROP LITE)检测DNA样品的纯度和浓度。
选用65对羊茅属高羊茅的基因组SSR分子标记的引物进行扩增分析[13]。选择形态差异大、海拔高差大的4份中华羊茅材料, 提取DNA, 利用65对高羊茅SSR 引物进行PCR扩增。选用多态性好、条带清晰的引物, 对全部24份中华羊茅材料进行分析。
SSR 反应体系(20 μ L):模板DNA 2 μ L (20 ng), SSR引物 2 μ L, 2× Es Taq MasterMix(Dye) 10 μ L, 加ddH2O 6 μ L。扩增反应在JY966 Thermal Cycler型PCR循环仪上进行。反应程序为:94 ℃预变性 5 min; 94 ℃变性30 s, 64 ℃退火 30 s, 72 ℃ 延伸50 s, 25个循环; 72 ℃ 延伸10 min, 4 ℃保存。扩增产物在6%聚丙烯酰胺凝胶电泳分离110 min, 电泳结束后进行银染并用数码相机照相保存。
根据PCR 扩增产物的电泳结果, 在相同迁移位置上, 将稳定、清晰的条带记为1, 无带或不易分辨的弱带记为0, 获得原始数据矩阵。统计其每对引物总扩增条带和多态位点、多态位点百分比(percent of polymorphism bands, PPB)和多态性信息含量(polymorphic information content, PIC)。在 NTSYS 软件计算供试材料间的Dice遗传相似系数, 并基于Dice遗传相似系数进行UPGMA聚类分析和主成分分析(principal component analysis, PCA)[14]。采用GenAlEx 6.5计算供试材料地理距离、海拔与遗传距离的Mantel相关性分析[15]。采用POPGENE 1.31计算不同海拔类群的Nei’ s基因多样性和香农指数(Shannon-Wiener index)[16]。
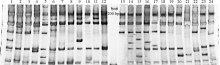
从65对高羊茅SSR引物中共筛选出17对条带型清晰、多态性好的引物。利用这些引物, 对24份中华羊茅的DNA进行扩增(表2)。本研究中供试材料的电泳条带均在100~500 bp(图 1)。同时不同引物扩增的位点数不同, 同一引物对24份资源扩增的条带类型和位点数也不一样。17对引物在供试的24份资源中共检测到102个位点, 其中多态性位点95个, 多态性比率为93.14%, 平均每对引物的多态性位点5.59个。每条引物扩增的位点数4~8个, 平均每条引物检测到6个位点, 多态性信息含量指数PIC值变化为0.240~0.470, 平均值为0.381。这也表明筛选的高羊茅SSR标记在中华羊茅中具有较好的通用性和高多态性。
本研究中24份中华羊茅种质资源间的遗传相似系数在0.188~0.675, 平均值为0.486。其中I-14-1-1与I-2-23-7之间的亲缘关系最远, 遗传相似系数为0.188; 09-124(青海平安)与09-141(青海平安)的亲缘关系最近, 遗传相似系数为0.675, 这都表明了供试的中华羊茅种质间具有丰富的遗传变异。
根据各个资源间的Dice遗传系数对24份中华羊茅种质资源进行UPGMA聚类, 结果表明在遗传距离0.52处可将其分为八大类(图2)。第Ⅰ 类由来自青海的1号(09-099)和来自西藏的9号(09-261)资源2份材料组成。第Ⅱ 类包括了5个材料, 均来自青海, 其中来自青海格尔木的3号(09-141)和来自青海平安的4号(09-124)材料具有非常近的亲缘关系。第Ⅲ 类由6份材料组成, 包括来自青海的5号、7号、11号、12号和来自西藏的6号, 以及来自四川的8号材料组成。第Ⅳ 类包括了6份材料, 其中3份来自青海, 2份来自西藏, 四川1份。第Ⅴ 类包括2份资源, 分别来自青海的23号(10-121)和西藏的24号(I-2-23-2)资源。第Ⅵ 类由1份材料组成, 是来自青海的10号(10-116)。第Ⅶ 类由来自四川的16号(I-14-1-1)材料构成。第VIII类由一份材料20号(I-2-23-7)来自青海的构成。这两份材料与其他材料间表现出了较远的亲缘关系, 在遗传上表现了一定的特异性。
 | 图2 24份中华羊茅种质资源基于遗传相似系数的UPGMA聚类图Fig. 2 UPGMA dendrogram of 24 Festuca sinensis germplasm resources based on genetic similarity coefficients |
基于供试材料间遗传相似系数进行PCA分析, 结果表明第1主成分的变异解释比率为62.01%, 第2主成分变异解释比率为3.40%, 第3主成分变异解释比率为3.24%, 3个主成分累计解释的变异比率为68.65%, 大体上能够反映中华羊茅各个资源间亲缘关系的远近, 与聚类的结果基本保持一致。
供试24份材料的遗传距离与地理距离的mantel检验结果表明二者之间相关性较低且不显著(r=0.062, P=0.139), 这与聚类分析的结果相一致。海拔与种质间的遗传距离之间的相关性也较低(r=0.05, P=0.492)。根据本研究中供试材料的海拔分布, 将供试材料分为3个不同的海拔类群, 即低海拔组(2 500-3 000 m)、中海拔组(3 000-3 600 m)和高海拔组(4 000-5 000 m)。不同海拔类群的遗传变异分析显示(表3), 分布于中度海拔(3 000-3 500 m)的中华羊茅种质具有最高的Nei’ s基因多样性(0.304 3)和香农多样性指数为0.462 2, 遗传变异较高; 高海拔地区(4 000-5 000 m)的种质遗传变异较低, 其Nei’ s基因多样性0.249 7, 香农多样性指数为0.395 6; 而低海拔类群种质(2 500-3 000 m)具有适中的遗传变异, 其Nei’ s基因多样性为0.284 9, 香农多样性指数为0.432 7。
| 表3 不同海拔类群材料的遗传变异 Table 3 Genetic variation of different elevation group materials |
由于不受环境影响, 不受限于植物的发育阶段, 分子标记已成为遗传多样性评价的强有力的主流研究手段[17, 18], 广泛应用于植物遗传育种的各个方面[19]。在羊茅属资源中, SSR也已经用于遗传评价、图谱构建、关联分析等工作[18]。Romina等[10]用15条引物检测161份来自不同国家的高羊茅(Festuca arundinacea), 共检测到214个多态性基因, 平均每条引物检测到5~24个, 遗传距离变幅范围为0.627~0.840, 体现了较高的遗传多样性。Saha等[13]在高羊茅中开发了152对SSR引物, 其中92%的引物能够扩增出条带。同时, 这些标记在不同的物种中表现出了不同的多态性比率。在羊茅属(Festuca)、黑麦草属(Lolium)等异花授粉植物中检测到66%的多态性标记, 而在水稻(Oryza sativa)和小麦(Triticum aestivum)中仅有43%和38%的多态性标记。本研究选用了其中的65对引物在中华羊茅中进行扩增, 最后筛选出了17对效果较好的标记。这也表明高羊茅引物在中华羊茅中具有较好的通用性和多态性, 是其资源遗传评价等有效的SSR标记来源。
基于遗传相似系数的聚类分析等结果表明, 供试材料的聚类并没有与其地理来源表现出较高的相关性, 其中西藏、四川的材料与青海的材料间聚类存在交叉。这可能与中华羊茅异花授粉的繁育方式相关, 不同地区种质间存在广泛的基因流, 如来自青海平安的7份材料除了3号(09-141)资源与4号(09-124)资源聚在一类, 其他5份资源分别与不同地理来源的资源聚在一类, 这与鸭茅(Dactylis glomerata)和多花黑麦草(L. multiflorum)等异花授粉牧草的种质资源评价特点表现出了一定的相似性[20, 21, 22, 23]。此外, 其中16号材料I-14-1-1和20号材料I-2-23-7在形态学和生理抗性上也表现出了特异性, 与其遗传特异性相吻合, 如16号叶片较为纤细, 对干旱、复水等处理非常敏感, 而20号材料则叶片较宽, 茎较粗壮有一定的耐旱性。
通过遗传距离与地理距离的分析发现两者没有明显的相关性, 但发现在海拔梯度呈现一定的遗传分化。严学兵等[23]发现海拔高度对披碱草(Elymus dahuricus)的遗传进化和选择起着巨大的作用; Chen等[24]对于青藏高原垂穗披碱草(E. nutans)遗传分化的研究也揭示了中度海拔的材料具有较高的遗传变异, 与本研究结果相似。本研究中高海拔种质具有相对较低的遗传变异, 这与其较高海拔具有相对较恶劣的生态环境有关, 也是其资源保护的重点; 而低海拔的遗传变异较低与人为的过度干扰等因素有关, 中等海拔具有相对较高的遗传多样性, 这些结果为中华羊茅种质资源的保护和利用提供了重要的参考。
本研究中采用SSR标记对来自青藏高原地区的24 份中华羊茅种质资源的遗传变异及亲缘关系进行了分析, 选用的高羊茅SSR标记在中华羊茅中表现出了较好的通用性, 结果揭示了供试材料间具有丰富的遗传变异, 明确了不同地理来源的材料间的遗传亲缘关系。其中I-14-1-1与其他野生种质表现出了较远的亲缘关系; 中等海拔3 000-3 600 m类群的材料具有较高的遗传变异, 研究结果为中华羊茅的资源保护及利用提供了重要参考。
(责任编辑 王芳)
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|